iGEM中的常微分方程模型
根据笔者去年参加iGEM的经验,整理得到本文,供后面参加iGEM并负责建模的同学参考。
本文中建模使用的编程语言是
MATLAB。
Logistic方程
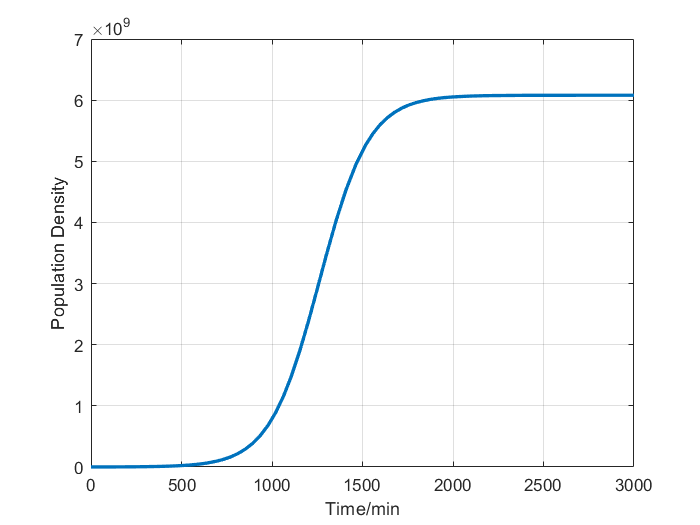
Logistic方程可以用于种群动力学模型的建模。设\(N\)是某种生物的种群密度,则 \[ \dfrac{\mathrm{d}N}{\mathrm{d}t}=rN\cdot\left(1-\dfrac{N}{K}\right), \] 其中\(r\)是生长率,\(K\)是环境容纳量。该方程可以求出解析解 \[ N(t)=\dfrac{KN_0\cdot \mathrm{e}^{rt}}{K+N_0\cdot(\mathrm{e}^{rt}-1)}, \] 其中\(N_0=N(0)\)是初始数量。该方程可以应用于:
- 给定生长率\(r\)和环境容纳量\(K\),预测种群数量的变化,需要使用
ode45函数。 - 给定种群数量随时间变化的数据,计算生长率并预测种群的最大数量,需要使用
lsqcurvefit函数。
物质守恒定律
所谓的物质守恒定律,说的是物质不会凭空消失,也不会凭空产生,只能由一种物质转化成另一种物质。在物质的转化过程中,就需要用到该定律。
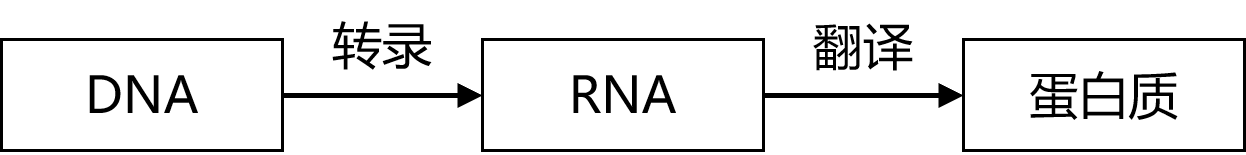
以中心法则为例,DNA首先转录为RNA,再由RNA翻译为蛋白质,这个过程就涉及到了物质的转化。
设蛋白质\(x\)的生产由启动子\(A\)决定,其浓度为\([x]\),对应的DNA浓度恒定且为\([dx]\),RNA浓度为\([rx]\)。对于转录的过程,有 \[
\dfrac{\mathrm{d}[rx]}{\mathrm{d}t}=k_{syn,x}\cdot\alpha_{A}\cdot[dx]-k_{de,x}\cdot[rx],
\] 其中\(k_{syn,x}\)是\(x\)对应的RNA转录速率,\(k_{de,x}\)是\(x\)对应的RNA降解速率,\(\alpha_{A}\)是启动子\(A\)的表达率,如果\(A\)受到其他因素的影响,可以将其写成\(\alpha_{A}=\alpha_{A,0}+\alpha_{A,i}\),其中\(\alpha_{A,0}\)是\(A\)的本底表达率,\(\alpha_{A,i}\)是\(A\)的诱导表达率。对于翻译的过程,有 \[
\dfrac{\mathrm{d}[x]}{\mathrm{d}t}=k_{psyn,x}\cdot [rx]-k_{pde,x}\cdot
[x],
\] 其中\(k_{psyn,x}\)是\(x\)的翻译速率,\(k_{pde,x}\)是\(x\)的降解速率。该定律可以应用于预测RNA和蛋白质的产量,需要对微分方程组使用ode45函数。
Michaelis-Menten方程
Michaelis-Menten方程是表示一个酶促反应的起始速度与底物浓度关系的速度方程。
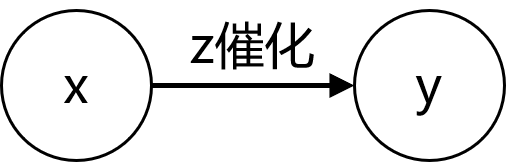
设蛋白质\(x\)转化成蛋白质\(y\)的过程由蛋白质\(z\)催化,并设它们的浓度分别为\([x]\)、\([y]\)和\([z]\),则有 \[
\dfrac{\mathrm{d}[y]}{\mathrm{d}t}=\dfrac{v_{max,z}\cdot[x]}{[x]+k_{m,z}},
\] 其中\(v_{max,z}=k_{cat,z}\cdot[z]\),\(k_{cat,z}\)是\(z\)的催化效率,\(k_{m,z}\)是常数。该方程可以用于预测蛋白质的产量,需要使用ode45函数。
建模主要的工作是模拟和预测,并根据结果来给出建议。要了解更多,可以参考2021年的建模结果:Team:XJTU-China/Model。



